|
Coautores
Patricia Dos Santos* Carmen Stanganelli** Irma Slavutsky***
Licenciada en Genética, Instituto de Medicina Experimental, CONICET-Academia Nacional de Medicina, CABA, Argentina*
Bioquímica, Instituto de Investigaciones Hematológicas, Academia Nacional de Medicina, CABA, Argentina**
Médica, Instituto de Medicina Experimental, CONICET-Academia Nacional de Medicina, CABA, Argentina***
|
|
Recepción del artículo: 3 de junio, 2019
|
|
Aprobación: 3 de junio, 2019
|
 Conclusión breve
Conclusión breve
La leucemia linfocítica crónica es la leucemia más frecuente en adultos de Occidente. Presenta un curso clínico altamente variable, con pacientes que requieren tratamiento inmediato y otros con un curso indolente de la enfermedad. Nuestro objetivo fue evaluar mutaciones de NOTCH1 en nuestros pacientes mediante ASO-PCR y secuenciación.
 Resumen
Resumen
La leucemia linfocítica crónica (LLC) es la leucemia más frecuente en adultos de Occidente. Presenta un curso clínico altamente variable, con pacientes que requieren tratamiento inmediato y otros con un curso indolente de la enfermedad. Los estudios genéticos constituyen herramientas de suma utilidad en esta enfermedad, encontrándose incorporados a las clasificaciones de riesgo internacionales. El análisis de los rearreglos genómicos y del estado mutacional de los genes IGHV (immunoglobulin heavy chain variable region) ha hecho factible establecer grupos de riesgo de alto valor pronóstico. Más recientemente, estudios de secuenciación de última generación permitieron la detección de mutaciones somáticas previamente desconocidas en esta afección, que podrían explicar la amplia variabilidad clínica observada en la LLC. Entre ellas, resultan de interés las observadas en el gen NOTCH1 (neurogenic locus notch homolog protein 1), cuya desregulación se asocia con el desarrollo tumoral. Estas mutaciones se acumulan en mayor medida en el exón 34 (80% de los casos) y en la región 3´UTR (untraslated region), lo que genera codones de terminación prematuros que originan una proteína NOTCH1 constitutivamente activa y más estable, los cuales se asocian con pronóstico adverso y refractariedad al tratamiento. Nuestro objetivo fue evaluar mutaciones de NOTCH1 en nuestros pacientes mediante ASO-PCR y secuenciación. Se detectaron mutaciones en el 4.4% de los casos, valor concordante con los datos internacionales (5% a 10%). Su inclusión en la caracterización genética de los pacientes con LLC permitirá refinar la categorización de los grupos de riesgo, aspecto de suma importancia tanto en el seguimiento clínico como en la toma de decisiones terapéuticas.
 Palabras clave
Palabras clave
gen NOTCH1, mutaciones, leucemia linfocítica crónica, citogenética, FISH, IgH
Clasificación en siicsalud
 Artículos originales> Expertos del Mundo>
Artículos originales> Expertos del Mundo>
página www.siicsalud.com/des/expertos.php/159835
Especialidades
 Principal: Genética Humana, Hematología,
Principal: Genética Humana, Hematología,
 Relacionadas: Diagnóstico por Laboratorio, Oncología,
Relacionadas: Diagnóstico por Laboratorio, Oncología,
 Enviar correspondencia a:
Enviar correspondencia a:
Irma Slavutsky, 1425, CABA, Argentina
NOTCH1 gene mutations in chronic lymphocytic leukemia
 Abstract
Abstract
Chronic lymphocytic leukemia (CLL) is the most common leukemia in the Western world. The disease has a highly variable clinical course, ranging from very indolent cases to patients with aggressive and rapidly progressing outcome. Genetic studies are useful tools in analyzing this pathology, and have been incorporated in international risk classifications. The analysis of genomic rearrangements and the mutational status of immunoglobulin heavy chain variable have allowed risk groups of high prognostic value to be established. More recently, next generation sequencing studies have identified novel somatic mutations that could explain the wide clinical variability of this pathology. Among them, the analysis of NOTCH1 (neurogenic locus notch homolog protein 1) gene mutations are of interest, as deregulation is associated with tumorigenesis.
NOTCH1 mutations are mostly located at exon 34 (80% of cases) and 3´UTR (untranslated region). They produce premature stop codons that produce a constitutively active and stable NOTCH1 protein. NOTCH1 mutations are associated with adverse prognosis and refractoriness to treatment. The aim of this study was to analyze NOTCH1 mutations in CLL patients by ASO-PCR and sequencing. Our results found 4.4% of cases with NOTCH1 mutated values concordant with international observations (5%-10%). Including them in the genetic status of CLL patients allows the characterization of risk groups, an aspect of great importance in clinical practice and therapeutic decisions, to be refined.
 Key words
Key words
NOTCH1 gene, mutations, chronic lymphocytic leukemia, cytogenetics, FISH, IgH
MUTACIONES DEL GEN NOTCH1 EN LA LEUCEMIA LINFOCÍTICA CRÓNICA
|
(especial para SIIC © Derechos reservados) |
|
Artículo completo
Leucemia linfocítica crónica
Las neoplasias del sistema linfoide constituyen un grupo heterogéneo de enfermedades, con un amplio espectro de variación desde el punto de vista clínico, morfológico, inmunofenotípico, citogenético y molecular. Entre ellas se encuentra la leucemia linfocítica crónica (LLC), que constituye la leucemia más frecuente en adultos de Occidente, dado que representa el 30% de los casos.1,2 Se caracteriza por la acumulación progresiva de linfocitos B maduros en sangre periférica, médula ósea y ganglios linfáticos, como resultado de un desequilibrio entre la disminución de la apoptosis y el aumento de la proliferación de un subgrupo de células leucémicas. Inicialmente se consideró que se trataba de una enfermedad estable originada por la simple acumulación de linfocitos B quiescentes como consecuencia de una disminución de la apoptosis, pero estudios posteriores demostraron la presencia de linfocitos tumorales con una alta capacidad proliferante,3 asociados con enfermedad más agresiva.4
Un aspecto característico de esta entidad es la diferencia en la evolución clínica de los pacientes, con algunos casos que requieren tratamiento inmediato y otros que presentan un curso más indolente de la enfermedad,5 sin que sean suficientes los sistemas de estadificación y los parámetros biológicos disponibles para predecir este comportamiento diferente. Esto hace necesario poder contar con marcadores que permitan clarificar en forma más precisa el curso clínico de los pacientes y su respuesta al tratamiento. En este aspecto, los estudios genéticos constituyen herramientas de suma utilidad en la comprensión de la heterogeneidad que la caracteriza, lo que resulta de relevancia por sus repercusiones clínicas en el diagnóstico, el pronóstico y la respuesta al tratamiento, encontrándose incorporados a las clasificaciones de riesgo internacionales. Estos estudios son de utilidad no solo como marcadores de valor diagnóstico o pronóstico, sino también en la comprensión de los mecanismos implicados en el proceso de desarrollo y progresión neoplásica.
En este aspecto, el análisis de los rearreglos genómicos y del estado mutacional de los genes IGHV (immunoglobulin heavy chain variable region) ha permitido establecer grupos de riesgo de alto valor pronóstico.6 La presencia de mutaciones a nivel de la región variable de la cadena pesada de las inmunoglobulinas, ubicada a nivel de 14q32, ha hecho factible dividir a la LLC en dos grupos de acuerdo con el nivel de homología respecto de la línea germinal: mutados (M) (< 98%), asociados con mejor pronóstico, y no mutados (NM) (= 98%), relacionados con peor evolución clínica.7-9 Por otra parte, los estudios citogenéticos han permitido detectar alteraciones clonales en un 40% a 50% de los casos, con repercusiones adversas en la evolución clínica de los pacientes.10,11 En tanto que la utilización de la técnica de FISH (fluorescence in situ hybridization), ha hecho factible detectar rearreglos genómicos en el 80% de los casos, así como el establecimiento de grupos de riesgo de valor pronóstico.12,13 En este aspecto encontramos la deleción monoalélica de 13q14, la anomalía más frecuente, observada en aproximadamente el 55% de los pacientes, asociada con buen pronóstico cuando se encuentra como única alteración; las deleciones de 11q22 (10%), donde se encuentra ubicado el gen ATM (ataxia telangiectasia mutated), y de 17p13 (5% a 8%), que involucra al gen TP53 (Tumor Protein P53), relacionadas con mala evolución clínica y escasa respuesta al tratamiento, así como la trisomía del cromosoma 12 (16%), asociada con un pronóstico intermedio.
Más recientemente, estudios de secuenciación de última generación (NGS; next generation sequencing) permitieron la detección de mutaciones somáticas previamente desconocidas en esta enfermedad, que podrían explicar, en parte, la amplia variabilidad clínica observada en estos casos y las respuestas disímiles a la terapia.14-16 Entre ellas cabe mencionar las observadas en el gen NOTCH1 (neurogenic locus notch homolog protein 1), que posee un papel crítico en funciones como proliferación, diferenciación y apoptosis, y cuya desregulación se asocia con el desarrollo tumoral.17,18
Características del gen NOTCH1
El gen NOTCH1 se encuentra ubicado a nivel de 9q34.3, y presenta 34 exones que abarcan 50 kb (Figura 1.A). Este gen codifica para un receptor transmembrana, que es sintetizado a partir de un único precursor y clivado por una convertasa en el aparato de Golgi. El receptor maduro posee un dominio extracelular (EC), conformado por 36 repeticiones del epidermal growth factor (EGF)-like-repeat, 3 repeticiones que poseen residuos de cisteína (LNR), un dominio de heterodimerización (HD) y una porción transmembrana (TM) seguida de un dominio intracelular (IC) (Figura 1.B). La activación de NOTCH1 ocurre cuando uno de sus dos ligandos, SERRATE/JAGGED o DELTA, expresados en una célula adyacente, se une al receptor. Dicha unión induce un cambio conformacional en TM, lo que permite un primer clivaje, el cual es realizado por la desintegrasa ADAM metalopeptidasa, que se encuentra localizada en el espacio extracelular, lo que provoca la liberación del dominio EC. Luego se produce un segundo clivaje por una gamma-secretasa, que determina la liberación del dominio IC, el cual es translocado al núcleo, donde desplaza al complejo CSL (CBF1/supresor de Hairless/Lag-1)-represores transcripcionales y se une al factor de transcripción RBP-Jk (human J kappa recombination signal sequence binding protein) para regular la expresión de varios genes blanco.17,19-21
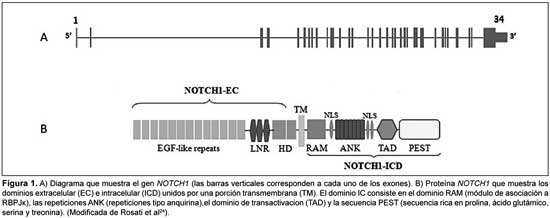
A la fecha se conocen solo algunos genes blanco de NOTCH1. Entre ellos encontramos dos familias: HES (Hairy enhancer of Split) y HEY (Hairy/enhancer of spit related with YRPW motif), cuyas proteínas son factores de transcripción helix-loop-helix que actúan como represores transcripcionales. También se conocen distintos genes blanco de la vía se señalización de NOTCH1, como ciclina D1, c-MYC, p21, p27, TK, mTOR y VEGE, entre otros, algunos de los cuales son dependientes de la señalización de NOTCH1 en varios tejidos, mientras que otros son específicos de tejido.22 Inicialmente, NOTCH1 fue considerado esencial para dirigir el compromiso de linaje de las células T a expensas del desarrollo de las células B, lo que demostró su potencial oncogénico en la leucemia de células T.23,24 Posteriormente, se encontró alterada la vía de NOTCH1 en numerosos tumores sólidos y neoplasias hematológicas.25 En un estudio pionero se demostró que las células de LLC expresan altos niveles del receptor NOTCH1 y de sus ligandos JAGGED1 y JAGGED2. En este aspecto, resulta de interés señalar que las células del estroma de los ganglios linfáticos también expresan JAGGED1, que induce la activación de NOTCH1, sustentando que una actividad aberrante de este gen podría contribuir a la aparición de la LLC.26 También se detectó que NOTCH1 podía estar constitutivamente activo, contribuyendo a la supervivencia de las células que lo expresan y confiriendo resistencia a la apoptosis.17
Mutaciones de NOTCH1 en la LLC
Las mutaciones de NOTCH1 se acumulan en mayor medida en el exón 34 y constituyen fundamentalmente eventos de corrimiento del marco de lectura y cambios sin sentido.24 La mutación más frecuente en los pacientes con LLC es una deleción de 2-pb (c.7541_7542het_delCT) en el exón 34 (80% de los casos), que afecta al extremo C-terminal, donde se encuentra localizado el dominio PEST (secuencia rica en prolina-glutamato-serina-treonina).27,28 En el año 2015, Puente y col.29 informaron una segunda mutación en la región 3´UTR (3'untranslated region). Las mutaciones más comunes de esta región no codificante ocurren principalmente en las posiciones 7668 371A>G y 7668 378A>G, y en forma menos frecuente en 7668 380A>C. Estas variantes crean un nuevo sitio de aceptación del corte y empalme (splicing), lo que favorece un evento de corte y empalme alternativo con un dador críptico en el exón 34.30 En ambos casos se genera la aparición de codones de terminación (stop) prematuros que originan una proteína NOTCH1 constitutivamente activa y más estable, que carece del dominio PEST en el extremo C-terminal, responsable de la degradación proteasomal.31,32 Esta activación de NOTCH1 aumenta, entre otros, la actividad del factor nuclear kappa B (NF-?B), cuya sobreexpresión tiene un papel crítico en la patogénesis de la LLC, promoviendo la proliferación y la supervivencia de las células leucémicas.33
NOTCH1 es el gen más comúnmente mutado en la LLC al momento del diagnóstico, y su frecuencia aumenta con la progresión de la enfermedad, por lo que es considerado un gen clave en la LLC, cuyas alteraciones probablemente representen un proceso oncogénico novedoso en esta entidad.24 Dichas mutaciones ocurren en un 5% a 10% de los pacientes al diagnóstico, con un incremento en la frecuencia que alcanza el 15% al 20% en los casos resistentes al tratamiento con fludarabina, llegando a aproximadamente el 30% en los casos que presentan transformación a síndrome de Richter,31,34-36 lo que indica una relación entre la desregulación de NOTCH1 y la respuesta al tratamiento. Asimismo, NOTCH1 mutado ha sido detectado en solo el 3% de los casos con linfocitosis B monoclonal,37 entidad asintomática que puede preceder la aparición de LLC. En estudios que emplearon NGS se ha observado la presencia de subclones con NOTCH1 mutado que se encuentran por debajo del límite de resolución de la técnica Sanger, lo que demuestra su papel en la progresión de la enfermedad y la refractariedad al tratamiento, lo que destaca la importancia de su detección.38,39 Asimismo, la detección reciente de activación no mutacional de NOTCH1 en LLC,40 sugiere una función más amplia de este probable marcador pronóstico en la enfermedad.
Las mutaciones de NOTCH1 se asocian con la presencia de trisomía del cromosoma 12 e IGHV-NM, lo cual genera mala evolución clínica con desregulación del ciclo celular y supervivencia breve de los pacientes,27,32,36,41 lo que sugeriría un papel crítico de la activación de dicho gen en estos pacientes.42 Asimismo, el análisis de la distribución de mutaciones en pacientes con receptores de células B de alta homología, denominados estereotipados, permitió detectar mayor frecuencia en los casos correspondientes a los subconjuntos #1, #6, #8 y #59 (22% a 34%), caracterizados por su agresividad clínica,43 lo que sugiere la función potencial de estos BCR estereotipados en la promoción o selección de mutaciones que influencian la evolución de la LLC.24
Considerando los diferentes regímenes de tratamiento, se observó peor evolución clínica en los casos tratados con una inducción de rituximab seguida de consolidación.44 Diferentes estudios demostraron menor tiempo al primer tratamiento y una supervivencia más corta en los pacientes que presentaban NOTCH1 mutado respecto de los tipos salvajes,31,45 resultados que fueron confirmados en estudios más recientes.46-48 Sin duda, la introducción de los nuevos regímenes de tratamiento que incluyen fármacos que actúan a nivel de las vías de señalización del BCR o de BCL2 ha beneficiado a estos pacientes, mejorando su respuesta al tratamiento.49,50 Asimismo, resulta de interés señalar que los casos con NOTCH1 mutado muestran baja expresión de CD20, resistencia a la inmunoterapia con anticuerpos anti-CD20 y pronóstico adverso,44,51 lo que sugiere la importancia de evaluar la presencia de estas mutaciones antes de la inclusión de pacientes en protocolos de tratamiento con este anticuerpo.
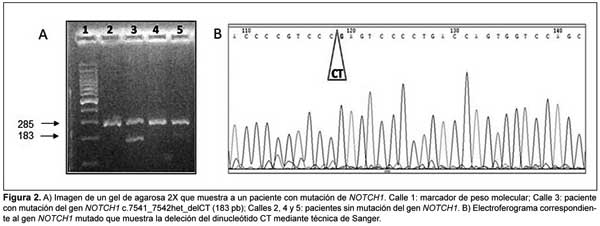
En cuanto a nuestra experiencia, al presente hemos analizado 90 pacientes con LLC (34 mujeres y 56 hombres; media de edad 65.4 años, rango: 44-82 años; con la siguiente distribución de estadios clínicos:52 0: 36.2%, I-II: 38.3% y III-IV: 25.5%). El estudio fue evaluado y aprobado por el Comité de Ética de los Institutos de la Academia Nacional de Medicina. Las muestras fueron tomadas con la conformidad y el consentimiento informado de los pacientes. En todos los casos se efectuó extracción de ADN genómico a partir de células mononucleares de sangre periférica, con el empleo de la técnica de ASO-PCR para la detección de la mutación c.7541_7542delCT. En una proporción de casos se realizó secuenciación bidireccional para confirmar la mutación. La reacción de PCR fue llevada a cabo con 3 primers distintos: uno inverso común Notch1_Rv (5'- AAGGCTTGGGAAAGGAAGC-3') y dos primers hacia adelante: Notch1_Fw C (5'-GTGACCGCAGCCCAGTT-3'), que amplifica tanto el alelo de tipo salvaje como el mutado, generando productos de 283 pb y 285 pb, respectivamente, y un primer Notch 1_Fw Mut (5'- TCCTCACCCCGTCCCGA-3'), que amplifica un segmento de 183 pb solo en presencia de la mutación.53. Asimismo, se efectuó en estos pacientes el estudio citogenético y citomolecular con el fin de evaluar la presencia de anomalías cromosómicas recurrentes en la LLC, así como el análisis del estado mutacional de IGHV, utilizando metodologías previamente descriptas.54,55
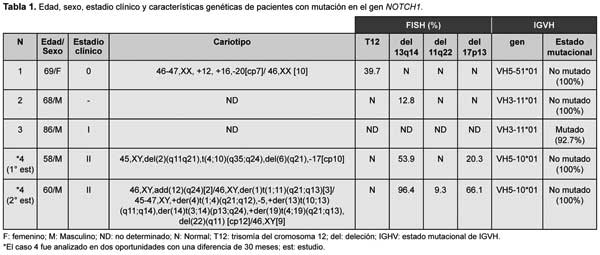
Nuestra cohorte mostró 4/90 (4.4%) casos con NOTCH1 mutado (Figuras 2.A y 2.B). Tres de estos pacientes presentaron IGHV-NM con 100% de homología con la línea germinal, y solo uno fue mutado con 92.7% de homología (Tabla 1). El caso 1 presentó un cariotipo complejo portador de las trisomías 12 y 16 y la pérdida del cromosoma 20. El caso 4 fue analizado en dos oportunidades, mostrando en ambos casos un cariotipo complejo, con múltiples anomalías estructurales y evolución clonal, tanto a nivel citogenético como citomolecular. Al momento de la primera muestra se observó que no poseía mutación en NOTCH1, que sí fue observada en el segundo estudio efectuado luego de 30 meses de evolución, lo que indica la presencia de evolución clonal también a nivel molecular. Este paciente presentó, además, mutación del gen TP53, asociación que ha sido descripta en el 1.2% al 2.6% de los casos.51,56
Sin duda, la incorporación del estudio de nuevas mutaciones, como NOTCH1, a la caracterización genética de los pacientes con LLC permitirá refinar la categorización de los grupos de riesgo citogenético, permitiendo la reclasificación de aproximadamente un 20% de casos de bajo riesgo. De acuerdo con el modelo integrado de alteraciones por FISH y mutaciones,57 los pacientes con NOTCH1 mutado pertenecen al grupo de riesgo intermedio, lo cual corresponde aproximadamente al 15% al 20% de los casos recién diagnosticados, con un 37% de supervivencia a 10 años. Esta posibilidad de recategorizar a los pacientes resulta de suma importancia en el seguimiento clínico, así como en la toma de decisiones terapéuticas, sobre todo teniendo en cuenta que las mutaciones de NOTCH1 son consideradas eventos tempranos en la aparición de la LLC.38 Dado que, a pesar de los avances a nivel terapéutico, la LLC continúa siendo una enfermedad incurable, la profundización de su caracterización genética es de suma importancia en el camino al desarrollo de nuevos agentes capaces de inhibir la actividad de NOTCH1,58 incrementando la actividad antitumoral sobre las células leucémicas.
 Bibliografía del artículo
Bibliografía del artículo
1. Chiorazzi N, Rai KR, Ferrarini M. Chronic lymphocytic leukemia. N Engl J Med 352:804-815, 2005.
2. Hallek M. Chronic lymphocytic leukemia: 2017 update on diagnosis, risk stratification, and treatment. Am J Hematol 92:946-965, 2017.
3. Damle RN, Calissano C, Chiorazzi N. Chronic lymphocytic leukaemia: a disease of activated monoclonal B cells. Best Pract Res Clin Haematol 23:33-45, 2010.
4. Palacios F, Abreu C, Prieto D, et al. Activation of the PI3K/AKT pathway by microRNA-22 results in CLL B-cell proliferation. Leukemia 29:115-25, 2015.
5. Dighiero D, Hamblin TJ. Chronic lymphocytic leukaemia. Lancet 371:1017-1029, 2008.
6. Gaidano G, Rossi D. The mutational landscape of chronic lymphocytic leukemia and its impact on prognosis and treatment. Hematology Am Soc Hematol Educ Program 2017:329-337, 2017.
7. Hamblin TJ, Davis Z, Gardiner A, Oscier DG, Stevenson FK. Unmutated Ig V(H) genes are associated with a more aggressive form of chronic lymphocytic leukemia. Blood 96:1848-1854, 1999.
8. Damle RN, Wasil T, Fais F, et al. Ig V gene mutation status and CD38 expression as novel prognostic indicators in chronic lymphocytic leukemia. Blood 94:1840-1847, 1999.
9. Stamatopoulos K, Belessi C, Hadzidimitriou A, et al. Immunoglobulin light chain repertoire in chronic lymphocytic leukemia. Blood 106:3575-3583, 2005.
10. Haferlach C, Dicker F, Schnittger S, Kern W, Haferlach T. Comprehensive genetic characterization of CLL: a study on 506 cases analysed with chromosome banding analysis, interphase FISH, IgV(H) status and immunophenotyping. Leukemia 21:2442-2451, 2007.
11. Travella A, Ripollés L, Aventin A, et al. Structural alterations in chronic lymphocytic leukaemia. Cytogenetic and FISH analysis. Hematol Oncol 31:79-87, 2013.
12. Döhner H, Stilgenbauer S, Benner A, et al. Genomic aberrations and survival in chronic lymphocytic leukemia. N Engl J Med 343:1910-1916, 2000.
13. Van Dyke DL, Werner L, Rassenti LZ, et al. The Dohner fluorescence in situ hybridization prognostic classification of chronic lymphocytic leukaemia (CLL): the CLL Research Consortium experience. Br J Haematol 173:105-113, 2016.
14. Gazzola A, Mannu C1, Rossi M, et al. The evolution of clonality testing in the diagnosis and monitoring of hematological malignancies. Ther Adv Hematol 5:35-47, 2014.
15. Baliakas P, Hadzidimitriou A, Sutton LA, et al. Recurrent mutations refine prognosis in chronic lymphocytic leukemia. Leukemia 29:329-336, 2015.
16. Malcikova J, Stano-Kozubik K, Tichy B, et al. Detailed analysis of therapy-driven clonal evolution of TP53 mutations in chronic lymphocytic leukemia. Leukemia 29:877-885, 2015.
17. Rosati E, Sabatini R, Rampino G, et al. Constitutively activated Notch signaling is involved in survival and apoptosis resistance of B-CLL cells. Blood 22:856-865, 2009.
18. Lobry C, Oh P, Aifantis I. Oncogenic and tumor suppressor functions of Notch in cancer: it's NOTCH what you think. J Exp Med 26:1931-1935, 2011.
19. Leong KG, Karsan A. Recent insights into the role of Notch signaling in tumorigenesis. Blood 107:2223-2233, 2006.
20. Hajdu M, Sebestyén A, Barna G, et al. Activity of the notch-signalling pathway in circulating human chronic lymphocytic leukaemia cells. Scand J Immunol 65:271-275, 2007.
21. Osborne BA. B-CLL kicks it up a Notch. Blood 22:765-766, 2009.
22. Liu N, Zhang J, Ji C. The emerging roles of Notch signaling in leukemia and stem cells. Biomark Res 1:23, 2013.
23. Rothenberg EV. T cell lineage commitment: identity and renunciation. J Immunol 186:6649-6655, 2011.
24. Rosati E, Baldoni S, De Falco F, et al. NOTCH1 Aberrations in Chronic Lymphocytic Leukemia. Front Oncol 8:229, 2018.
25. Di Ianni M, Baldoni S, Del Papa B, et al. NOTCH1 Is Aberrantly Activated in Chronic Lymphocytic Leukemia Hematopoietic Stem Cells. Front Oncol 8:105, 2018.
26. Arruga F, Gizdic B, Serra S, et al. Functional impact of NOTCH1 mutations in chronic lymphocytic leukemia. Leukemia 28:1060-1070, 2014.
27. Rossi D, Rasi S, Fabbri G, et al. Mutations of NOTCH1 are an independent predictor of survival in chronic lymphocytic leukemia. Blood 119:521-529, 2012.
28. Gianfelici V. Activation of the NOTCH1 pathway in chronic lymphocytic leukemia. Haematologica 97:328-330, 2012.
29. Puente XS, Beà S, Valdés-Mas R, et al. Non-coding recurrent mutations in chronic lymphocytic leukaemia. Nature 526:519-524, 2015.
30. Abramenko IV, Bilous NI, Chumak AA, Dyagil IS, Martina ZV. Analysis of the 3'UTR region of the NOTCH1 gene in chronic lymphocytic leukemia patients. Exp Oncol 40:211-217, 2018.
31. Fabbri G, Rasi S, Rossi D, et al. Analysis of the chronic lymphocytic leukemia coding genome: role of NOTCH1 mutational activation. J Exp Med 208:1389-1401, 2011.
32. Zou Y, Fan L, Xia Y, et al. NOTCH1 mutation and its prognostic significance in Chinese chronic lymphocytic leukemia: a retrospective study of 317 cases. Cancer Med 7:1689-1696, 2018.
33. Xu ZS, Zhang JS, Zhang JY, et al. Constitutive activation of NF-? signaling by NOTCH1 mutations in chronic lymphocytic leukemia. Oncol Rep 33:1609-1614, 2015.
34. Albi E, Baldoni S, Aureli P, et al. Ibrutinib treatment of a patient with relapsing chronic lymphocytic leukemia and sustained remission of Richter syndrome. Tumori 103:37-40, 2017.
35. Rossi D, Spina V, Forconi F, et al. Molecular history of Richter syndrome: origin from a cell already present at the time of chronic lymphocytic leukemia diagnosis. Int J Cancer 130:3006-3010, 2011.
36. Del Giudice I, Rossi D, Chiaretti S, et al. NOTCH1 mutations in 12 chronic lymphocytic leukemia (CLL) confer an unfavorable prognosis, induce a distinctive transcriptional profiling and refine the intermediate prognosis of 12 CLL. Haematologica 97:437-441, 2012.
37. Rasi S, Monti S, Spina V, Foà R, Gaidano G, Rossi D. NOTCH1 mutations in monoclonal B-cell lymphocytosis. Hematologica 97:153-154, 2012.
38. Nadeu F, Delgado J, Royo C, et al. Clinical impact of clonal and subclonal TP53, SF3B1, BIRC3, NOTCH1, and ATM mutations in chronic lymphocytic leukemia. Blood 127:2122-2130, 2016.
39. Rasi S, Khiabanian H, Ciardullo C, et al. Clinical impact of small subclones harboring NOTCH1, SF3B1 or BIRC3 mutations in chronic lymphocytic leukemia. Haematologica 101:135-138, 2016.
40. Fabbri G, Holmes AB, Viganotti M, et al. Common nonmutational NOTCH1 activation in chronic lymphocytic leukemia. Proc Natl Acad Sci USA 114:2911-2919, 2017.
41. Jeromin S, Weissmann S, Haferlach C, et al. SF3B1 mutations correlated to cytogenetics and mutations in NOTCH1, FBXW7, MYD88, XPO1 and TP53 in 1160 untreated CLL patients. Leukemia 18:108-117, 2014.
42. Balatti V, Bottoni A, Palamarchuk A, et al. NOTCH1 mutations in CLL associated with trisomy 12. Blood 119:329-331, 2012.
43. Sutton LA, Young E, Baliakas P, et al. Different spectra of recurrent gene mutations in subsets of chronic lymphocytic leukemia harboring stereotyped B-cell receptors. Hematologica 101:959-967, 2016.
44. Dal Bo M, Del Principe MI, Pozzo F, et al. NOTCH1 mutations identify a chronic lymphocytic leukemia patient subset with worse prognosis in the setting of a rituximab-based induction and consolidation treatment. Ann Hematol 93:1765-1774, 2014.
45. Sportoletti P, Baldoni S, Cavalli L, et al. NOTCH1 PEST domain mutation is an adverse prognostic factor in B-CLL. Br J Haematol 151:404-406, 2010.
46. Sportoletti P, Baldoni S, Del Papa B, et al. A revised NOTCH1 mutation frequency still impacts survival while the allele burden predicts early progression in chronic lymphocytic leukemia. Leukemia 28:436-439, 2014.
47. Guièze R, Robbe P, Clifford R, et al. Presence of multiple recurrent mutations confers poor trial outcome of relapsed/refractory CLL. Blood 126:2110-2117, 2015.
48. Putowski M, Podgórniak M, Piróg M, et al. Prognostic impact of NOTCH1, MYD88, and SF3B1 mutations in Polish patients with chronic lymphocytic leukemia. Pol Arch Intern Med 127:238-244, 2017.
49. Del Poeta G, Del Principe MI, Postorino M, et al. Apoptosis resistance and NOTCH1 mutations impair clinical outcome in chronic lymphocytic leukemia (CLL) patients treated with ibrutinib. Blood 130(Suppl 1):261, 2017.
50. Brown JR, Hillmen P, O'Brien S, et al. Extended follow-up and impact of high-risk prognostic factors from the phase 3 RESONATE study in patients with previously treated CLL/SLL. Leukemia 32:83-91, 2018.
51. Stilgenbauer S, Schnaiter A, Paschka P, et al. Gene mutations and treatment outcome in chronic lymphocytic leukemia: results from the CLL8 trial. Blood 123:3247-3254, 2014.
52. Rai KR, Sawitsky A. A review of the prognostic role of cytogenetic, phenotypic, morphologic, and immune function characteristics in chronic lymphocytic leukemia. Blood Cells 12:327-38, 1987.
53. Lionetti M, Fabris S, Cutrona G, et al. High-throughput sequencing for the identification of NOTCH1 mutations in early stage chronic lymphocytic leukaemia: biological and clinical implications. Br J Haematol 165:629-639, 2014.
54. Palmitelli M, Stanganelli C, Stella F, et al. Analysis of basal chromosome instability in patients with chronic lymphocytic leukaemia. Mutagenesis 2019. doi: 10.1093/mutage/gez009.
55. Stanganelli C, Travella A, Bezares R, Slavutski I. Immunoglobulin gene rearrangements and mutational status in Argentinean patients with chronic lymphocytic leukemia. Clin Lymph, Myeloma Leuk 13:447-457, 2013.
56. Weissmann S, Roller A, Jeromin S, et al. Prognostic impact and landscape of NOTCH1 mutations in chronic lymphocytic leukemia (CLL): a study on 852 patients. Leukemia 27:2393-2396, 2013.
57. Rossi D, Rasi S, Spina V, et al. Integrated mutational and cytogenetic analysis identifies new prognostic subgroups in chronic lymphocytic leukemia. Blood 121:1403-1412, 2013.
58. Baldoni S, Del Papa B, Dorillo E, et al. Bepridil exhibits anti-leukemic activity associated with NOTCH1 pathway inhibition in chronic lymphocytic leukemia. Int J Cancer 143:958-970, 2018.
|
|
©
Está
expresamente prohibida la redistribución y la redifusión de todo o parte de los
contenidos de la Sociedad Iberoamericana de Información Científica (SIIC) S.A. sin
previo y expreso consentimiento de SIIC |

|
|



