|
Coautores
Ricardo Tristão Sá* Thaís Tristão Tovar** Marcelo dos Santos*** Franciane Figueiredo da Silva**** Iuri Drumond Louro*****
Médico, Scola Superior de Ciências da Santa Casa de Misericórdia de Vitória, Vitória, Brasil*
Biologo, Universidade Federal do Espírito Santo, Vitória, Brasil**
Biólogo, Universidade Federal do Rio Grande do Norte, Natal, Brasil***
Especialista en estadística, Universidade Federal do Espírito Santo, Vitória, Brasil****
Médico, Universidade Federal do Espírito Santo, Vitória, Brasil*****
|
|
Recepción del artículo: 0 de , 0000
|
|
Aprobación: 0 de , 0000
|
 Conclusión breve
Conclusión breve
Este é o primeiro estudo para determinar as frequências dos SNPs -590C/T (IL-4 gene), -174G/C (IL-6 gene) e 874A/T (IFN-gamma gene) nas populações geral e de origem pomerana do Espirito Santo, Brasil. A caracterização de duas populações distintas no mesmo estado nos permite compreender as especificidades encontradas na mesma região geográfica e nos ajudará a estabelecer estratégias de prevenção e controle específicos para cada população.
 Resumen
Resumen
A caracterização das diferentes populações serve como referência para estudos de associação de polimorfismos de nucleotídeo único com características de doenças infecciosas, por exemplo. Os estudos de associação podem representar uma ferramenta valiosa para a compreensão de padrões de resposta imunológica, auxiliando na identificação de grupos de risco para o desenvolvimento de infecções graves, bem como no desenvolvimento de terapias e vacinas mais eficazes. Polimorfismos de nucleotídeo único em genes de citocinas influenciam o resultado de muitas doenças infecciosas. Nesse trabalho, analisamos a frequência dos polimorfismos de nucleotídeo único denominados interleucina-4: -590C/T (rs2243250), interleucina-6: -174G/C (rs1800795) e interferon-gama: 874A/T (rs2430561). As análises foram realizadas na população geral do estado do Espírito Santo, Brasil, bem como na população isolada de origem pomerana, no mesmo estado. O DNA genômico periférico foi extraído de cem indivíduos da população geral e de 59 pomeranos. A identificação da variante polimórfica foi realizada por reação em cadeia da polimerase seguida da reação de polimofismo de comprimento de fragmento de restrição. Polimorfismos de nucleotídeo único estiveram em equilíbrio de Hardy-Weinberg em ambas as populações, exceto o -174G/C na população geral (p = 0,00). Observou-se uma diferença estatisticamente significativa nas distribuições genotípicas dos polimorfismos no genes da interleucina-6 (p = 0,03) e do interferon-gama (p = 0,07) entre as duas populações. A distância genética entre as duas populações não foi estatisticamente significativa (FST > 0,05). Este é o primeiro estudo a determinar as frequências desses polimorfismos de nucleotídeo único nas populações geral e pomerana do Espirito Santo, Brasil.
 Palabras clave
Palabras clave
interleucina-4, interleucina-6, polimorfismos, Espírito Santo-Brasil, interferon-gama
Clasificación en siicsalud
 Artículos originales> Expertos del Mundo>
Artículos originales> Expertos del Mundo>
página www.siicsalud.com/des/expertos.php/148174
Especialidades
 Principal: Genética Humana, Inmunología,
Principal: Genética Humana, Inmunología,
 Relacionadas: Epidemiología, Infectología,
Relacionadas: Epidemiología, Infectología,
 Enviar correspondencia a:
Enviar correspondencia a:
Raquel Spinassé Raquel, 29075-910, Vitória, Brasil
 Abstract
Abstract
The characterization of different populations serves as reference for association studies of single nucleotide polymorphisms with characteristics of infectious disease, specifically. Single nucleotide polymorphisms in cytokine genes influence the outcome of many infectious diseases. We have analyzed the frequency of single nucleotide polymorphism interleukin-4 -590C/T (rs2243250), interleukin-6 -174G/C (rs1800795) and interferon-gamma +874A/T (rs2430561). Analyses were performed in the general population of the Espirito Santo State, Brazil, as well as in the Pomeranian isolate population, in the same state. Peripheral genomic DNA was extracted from one hundred healthy individuals of the General population and from 59 Pomeranians. Polymorphic variant identification was performed by polymerase chain reaction-restriction fragment length polymorphism. Single nucleotide polymorphisms were in Hardy-Weinberg equilibrium in both populations, except the -174G/C in the general population (p = 0.00). We observed a statistically significant difference in interleukin-6 (p = 0.03) and interferon-gamma (p = 0.07) single nucleotide polymorphisms distributions between the two populations, allowing for the determination of distinct cytokine single nucleotide polymorphisms profiles. The genetic distance between the two populations was not statistically significant (Fst>0.05). This is the first study to determine the frequencies of these single nucleotide polymorphisms in the general and Pomeranian populations of ES, Brazil.
 Key words
Key words
interleukin-4, interleukin-6, polymorphisms, Espirito Santo-Brazil, interferon-gamma
ANÁLISE DE POLIMORFISMOS EM GENES QUE CODIFICAM CITOCINAS EM DUAS POPULAÇÕES DO ESPÍRITO SANTO, BRASIL
|
(especial para SIIC © Derechos reservados) |
|
Artículo completo
Introdução
Na maioria das doenças infecciosas, a resposta imune adquirida naturalmente resulta na eliminação do agente infeccioso ou em uma resposta persistente mediada por proteínas chamadas citocinas, as quais são segregadas durante as respostas imunológicas inata e adaptativa.1,2 O resultado de uma infecção depende de um equilíbrio entre as citocinas pró e anti-inflamatórias.2,3
As variantes alélicas, tambpem denominadas polimorfismos de nucleotídeo único (SNPs) em genes que codificam citocinas têm sido estudadas por influenciarem a transcrição gênica e a patogênese de várias doenças infecciosas.2,4-6 SNPs em vários genes que codificam citocinas conhecidas têm sido considerados importantes fatores do hospedeiro a influenciar a resposta à infecção e o prognóstico clínico.3 Entre as citocinas anti-inflamatórias mais estudados está a interleucina-4 (IL-4), e entre as mais importantes citocinas pró-inflamatórias estão a Interleucina-6 (IL-6) e o interferon-gama (INF-?).
A IL-4 é uma citoquina anti-inflamatória que promove respostas dos linfócitos T helper tipo 2 (Th2).7,8 O gene IL-4 está localizado na região 5q31.1. A substituição de uma citosina (C) por uma timina (T) na região promotora do gene, SNP -590C/T (2.243.250), pode aumentar a produção de IL-4,9 alterando a resposta imune contra várias infecções.10
A IL-6 é uma citocina pró-inflamatória envolvida na resposta de fase aguda, na maturação de células B, na diferenciação de macrófagos e na diferencição de células T helper tipo 1 (Th1)/Th2 (IL-6 promove a diferenciação de Th2 e inibe, simultaneamente, a diferenciação de Th1).11 O gene IL-6 é mapeado na região 7p21.24 e acredita-se que a presença de guanina (G) no seu promotor, SNP -174G/C (1.800.795), está associada com níveis plasmáticos superiores de IL-6. Este fato foi estudado em várias doenças infecciosas.12,13
O INF-? é uma citocina pró-inflamatória segregada principalmente pelas células Th1 CD4, Th1 CD8 e células natural killer (NK).14 O papel principal desta citocina é induzir funções microbicidas mediadas por macrófagos15 e promover a defesa do hospedeiro contra patógenos intracelulares.14 O gene INF-? está localizado na região 12q24.1 e o seu SNP 874T/A (2.430.561) tem sido largamente estudado em doenças infecciosas.14 O alelo T correlaciona-se com a expressão elevada de IFN-?, ao passo que o alelo com adenina (A) correlaciona-se com baixa expressão de INF-?.17-19
A região serrana do Espírito Santo (ES) possui a terceira maior população de ascendência pomerana. Esses descendentes emigraram originalmente do norte da Europa e, por causa de seu casamento preferencial por hábitos religiosos e culturais, apresentam pouca ou nenhuma mistura reprodutiva com outras populações locais,20,21 sugerindo um considerável grau de endogamia.
A caracterização populacional do perfil de citocinas pode representar uma ferramenta valiosa para a compreensão de padrões de resposta imunológica, auxiliando na identificação de grupos de risco para o desenvolvimento de infecções graves, bem como no desenvolvimento de terapias e vacinas mais eficazes. A caracterização de duas populações distintas no mesmo estado nos permite compreender as especificidades encontradas em cada região geográfica e nos ajuda a estabelecer estratégias de prevenção e controle específicos para cada população.
Materiais e métodos
Este estudo foi aprovado pelo Comitê de Ética e Pesquisa da Universidade Federal do ES e todos os pacientes assinaram o termo de consentimento (protocolo nº 190/11).
Amostra
Cinco mililitros (ml) de sangue periférico foram colhidos de 100 voluntários saudáveis ??da população geral do ES e 3-5 gotas de sangue periférico foram coletadas em FTA® Elute Cards (Whatman, EUA) de 59 voluntários saudáveis ??da população de origem pomerana de Santa Maria de Jetibá, ES.
Genotipagem dos SNPs
O DNA genômico foi isolado com uso da extração com fenol/clorofórmio ou seguindo as recomendações do fabricante do cartão FTA® Elute (Whatman, EUA). Os alelos dos genes INF-?, IL-4 e IL-6 foram detectados por reação em cadeia da polimerase seguida da reação de polimorfismo de comprimento de fragmento de restrição (PCR-RFLP). As sequências dos Primers, as enzimas de restrição e as condições das reações são mostradas na Tabela 1.
Tabla 1
A análise estatística
As frequências genotípicas foram testadas para o equilíbrio de Hardy-Weinberg (HWE). Os testes do qui-quadrado (x2) e exato de Fisher foram usados ??para análisae a diferença entre as populações e a confirmação foi obtida pelo teste Lilliefors (a significância estatística foi considerada quando p < 0.05). O modelo F-estatística foi utilizado para avaliar a distãncia genética entre as populações. Os cálculos estatísticos foram realizados no Epi Info® v3.4.3, 2007 e Statsoft Statistica® software v7.0.61.0.
Resultados
Os resultados da genotipagem são representados nas Figuras 1, 2 e 3. As frequências alélicas e genotípicas dos SNPs são apresentados na Tabela 2. As frequências genotípicas foram significativamente diferentes entre as duas populações para os genes INF -? (p = 0.007) e IL-6 (p = 0.03) (Tabela 2). Todos os polimorfismos estiveram em HWE em ambas as populações, excepto o SNP no gene IL-6, na população geral (p = 0.00) (Tabela 2). O coeficiente da distância genética (FST), uma medida da diferenciação de populações a partir de polimorfismos genéticos, é descrito na Tabela 2. Os resultados não mostraram nenhuma distância genética entre as duas populações (p> 0.05).
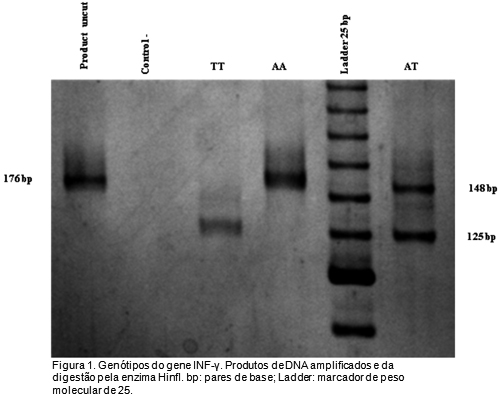
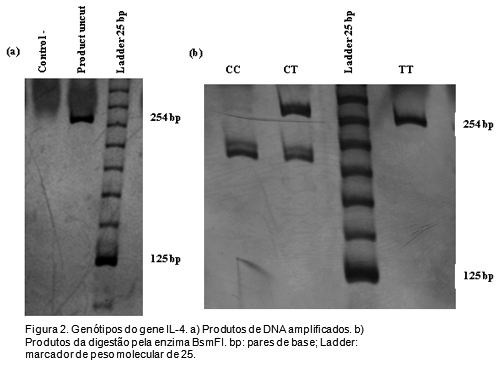
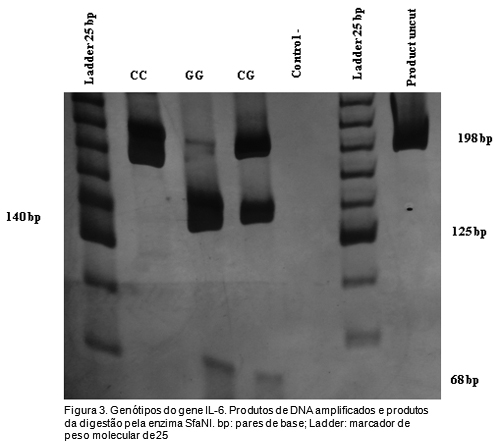
Tabla 2
Discussão
Este estudo caracterizou as populações geral e de origem pomerana do ES, Brasil quanto a 3 SNPs em genes que codificam citocinas: uma citocina anti-inflamatória (IL-4) e duas citocinas pró-inflamatórias (IL-6 e IFN-y). Estas citocinas desempenham papéis importantes no resultado de várias doenças infecciosas, por participarem da resposta imune do hospedeiro a patógenos, de processos inflamatórios, dentre outras funcões.7 Pelo fato das frequências alélicas e genotípicas de SNPs serem específicas para cada população, 7,12,13,22-24 estudos de caracterização populacional podem ajudar a determinar os perfis genéticos específicos, bem como a resposta regional específica para as doenças infecciosas: resistência ou susceptibilidade do hospedeiro; gravidade e permanência do sintomas clínicos. A caracterização das diferentes populações serve como referência para estudos de associação de SNPs com características de doenças infecciosas, especificamente. Os estudos de associação são importantes para melhorar o conhecimento da imunologia de doenças infecciosas e assim, de formas de tratamento e prevenção.
A distribuição genotípica dos SNPs nos genes IL-6 e IFN-y foi significativamente diferente nas duas populações. Ao contrário do que foi observado em Dettogni e col.25 e Stur e col.,26 neste estudo houve uma diferença de frequências genotípicas entre as duas populações do ES. Esta observação nos leva a concluir que, embora haja fluxo gênico entre as duas populações, alguns SNPs permanecem com frequências diferentes. Isto sugere que estas duas populações ainda podem ter algum grau de isolamento. No entanto, os valores Fst demonstram que a distância genética entre as duas populações não é estatisticamente significativa, com valores > 0.05, sugerindo mistura das duas populações em questão. São necessários mais estudos com amostras maiores para resulatdos mais eclarecedores.
O SNP no gene IL-6 não esteve em HWE na população geral, mas como em outros estudos,23,24 o genótipo mais frequente foi o homozigoto GG. Tem sido relatado que muitos SNPs podem não estar em HWE por razões não óbvias, especialmente se a população for etnicamente diversa.27 Provavelmente, a grande mistura genética da população brasileira e, consequentemente, da população geral do ES, pode ter influenciado nossos resultados. Dos Santos e col.28 sugerem que a população brasileira está sob um processo de miscigenação, o que poderia explicar o fato de alguns SNPs não estarem em HWE.
Este é o primeiro estudo a determinar as frequências dos SNPs -590C/T (IL-4), -174G/C (IL-6) e 874A/T (IFN-?) nas populações gerais e Pomeranian do ES, Brasil.
 Bibliografía del artículo
Bibliografía del artículo
1. Marty AM, Jahrling PB, Geisbert TW. Viral hemorrhagic fevers. Clin Lab Med 26:345-386, 2006.
2. Medina TS, Costa SP, Oliveira MD, Ventura AM, Souza JM, Gomes TF, et al. Increased interleukin-10 and interferon-gamma levels in Plasmodium vivax malaria suggest a reciprocal regulation which is not altered by IL-10 gene promoter polymorphism. Malar J 10:264, 2011.
3. Lee JY, Kim HY, Kim KH, Kim SM, Jang MK, Park JY, et al. Association of polymorphism of IL-10 and TNF-A genes with gastric cancer in Korea. Cancer Lett 225:207-214, 2005.
4. Bidwell J, Keen L, Gallagher G, Kimberly R, Huizinga T, McDermott MF, et al. Cytokine gene polymorphism in human disease: on-line databases. Genes Immun 1:3-19, 1999.
5. Hurme M, Lahdenpohja N, Santtila S. Gene polymorphisms of interleukins 1 and 10 in infectious and autoimmune diseases. Ann Med 30:469-473, 1998.
6. Rad R, Dossumbekova A, Neu B, Lang R, Bauer S, Saur D, et al. Cytokine gene polymorphisms influence mucosal cytokine expression, gastric inflammation, and host specific colonisation during Helicobacter pylori infection. Gut 53:1082-1089, 2004.
7. Dittmer U, Peterson KE, Messer R, Stromnes IM, Race B, Hasenkrug KJ. Stromnes IM, Race B, Hasenkrug KJ. Role of interleukin-4 (IL-4), IL-12, and gamma interferon in primary and vaccine-primed immune responses to Friend retrovirus infection. J Virol 75:654-660, 2001.
8. Liblau RS, Singer SM, McDevitt HO. Th1 and Th2 CD4 T cells in the pathogenesis of organ-specific autoimmune diseases. Immunol Today 16:34-38, 1995.
9. Chu H, Wang M, Yan F, Zhong D, Shi D, Ma L, et al. Polymorphisms in the IL-13 and IL-4R genes are associated with the development of renal cell carcinoma. Ann Oncol 23:2114-2121, 2012.
10. Moran TM, Isobe H, Fernandez-Sesma A, Schulman JL. Interleukin-4 causes delayed virus clearance in influenza virus-infected mice. Journal of Virology 70:5230-5235, 1996.
11. Diehl S, Rincon M. The two faces of IL-6 on Th1/Th2 differentiation. Mol Immunol 39:531-536, 2001.
12. Fernandez-Real JM, Vendrell J, Richart C, Gutierrez C, Ricart W. Platelet count and interleukin 6 gene polymorphism in healthy subjects. BMC Med Genet 2:6, 2001.
13. Honsawek S, Deepaisarnsakul B, Tanavalee A, Yuktanandana P, Bumrungpanichthaworn P, Malila S, Saetan N. Association of the IL-6 -174G/C gene polymorphism with knee osteoarthritis in a Thai population. Genet Mol Res 10:1674-1680, 2011.
14. Cardoso CC, Pereira AC, Brito de- Souza VN, Dias Baptista IM, Maniero VC, Venturini J, et al. IFNG 874 T>A single nucleotide polymorphism is associated with leprosy among Brazilians. Hum Genet 128:481-490, 2010.
15. Nathan CF, Prendergast TJ, Wiebe ME, Stanley ER, Platzer E, Remold HG, et al. Activation of human macrophages. Comparison of other cytokines with interferon-gamma. J Exp Med 160:600-605, 1984.
16. Kimkong I, Nakkuntod J, Sodsai P, Hirankarn N, Kitkumthorn N. Association of interferon-gamma gene polymorphisms with susceptibility to oral lichen planus in the Thai population. Arch Oral Biol 57:491-494, 2012.
17. Lopez-Maderuelo D, Arnalich F, Serantes R, González A, Codoceo R, Madero R, et al. Interferon-gamma and interleukin-10 gene polymorphisms in pulmonary tuberculosis. Am J Respir Crit Care Med 167:970-975, 2003.
18. Murray CJ, Styblo K, Rouillon A. Tuberculosis in developing countries: burden, intervention and cost. Bull Int Union Tuberc Lung Dis 65:2-20, 1990.
19. Vallinoto AC, Graça ES, Araujo MS, Azevedo VN, Cayres-Vallinoto I, Machado LFA, et al. IFNG 874T/A polymorphism and cytokine plasma levels are associated with susceptibility to Mycobacterium tuberculosis infection and clinical manifestation of tuberculosis. Hum Immunol 71:692-696, 2010.
20. Bahia J. A lei da vida: confirmacção, evasão escolar e reinvenção da identidade entre os pomeranos. Educ Pesqui 27:69-82, 2001.
21. Domingues RB, Aquino CCH, Santos JG, Silva ALP, Kuster GW. Prevalence and impact of headache and migraine among Pomeranians in Espirito Santo, Brazil. Arq Neuropsiquiatr 64:954-957, 2006.
22. Ben Selma W, Harizi H, Bougmiza I, Hannachi N, Ben Kahla I, Zaineni R, et al. Interferon gamma 874T/A polymorphism is associated with susceptibility to active pulmonary tuberculosis development in Tunisian patients. DNA Cell Biol 30:379-387, 2011.
23. Moreira ST, Cardoso DM, Visentainer JE, Fonsar UJV, Moliterno RA. The possible protective role of the Il6-174GC genotype in dengue fever. Open Trop Med J 1:87-91, 2008.
24. Pieroni F, Lourenço DM, Morelli VM, Maffei FH, Zago MA, Franco RF. Cytokine gene variants and venous thrombotic risk in the BRATROS (Brazilian Thrombosis Study). Thromb Res 120:221-229, 2007.
25. Dettogni RS, Tristão-Sa R, Tovar TT, Louro ID. Polymorphic genetic variation in immune system genes: a study of two populations of Espirito Santo, Brazil. Mol Biol Rep 40:4843-4849, 2013.
26. Stur E, Silveira AN, Selvatici LS, Alvez LNR, Wolfgramm EV, Tovar TT, et al. Polymorphism analysis of MTHFR, factor II, and factor V genes in the Pomeranian population of Espirito Santo, Brazil. Genet Test Mol Biomarkers 16:219-222, 2012.
27. Weinberg CR, Morris RW. Invited commentary: Testing for Hardy-Weinberg disequilibrium using a genome single-nucleotide polymorphism scan based on cases only. Am J Epidem 158:401-403, 2003.
28. Dos Santos M, Stur E, Maia LL, Agostini LP, Peterle GT, Mendes SO, et al. Genetic variability of inflammatory genes in the Brazilian population. Genet Test Mol Biomarkers 17:844-848, 2012.
|
|
©
Está
expresamente prohibida la redistribución y la redifusión de todo o parte de los
contenidos de la Sociedad Iberoamericana de Información Científica (SIIC) S.A. sin
previo y expreso consentimiento de SIIC |

|
|




